Blog
Librerías usadas
Para realizar consultas SQL se utiliza el paquete sqldf (CRAN - Readme, Docs, Github) y para la presentación de tablas y gráficos se utilizan knitr, summarytools, DT, ggplot2, gridExtra, grid, ggpubr y lattice. Una vez instalados se deben cargar mediante library().
library('sqldf')
library('knitr')
library('summarytools')
library('DT')
library('ggplot2')
library('gridExtra')
library('grid')
library('ggpubr')
library('lattice')Datos de diabetes
Estos datos representan un subconjunto de los utilizados en Tibshirani et al. (2004)
Hay 10 variables, que incluyen edad, sexo, índice de masa corporal (IMC) y presión arterial media (MAP) de 442 pacientes, así como seis mediciones de suero sanguíneo (tc, ldl, hdl, tch, ltg y glu). La respuesta (Y) es una medida continua de la progresión de la enfermedad un año después de las mediciones iniciales.
Importamos los datos a R y cambiamos los nombres de las columnas para que sean más amigables:
db <- read.delim('https://www4.stat.ncsu.edu/~boos/var.select/diabetes.tab.txt')
colnames(db) <- c("Edad", "Sexo", "IMC", "PAM", "tc", "ldl", "hdl", "tch", "ltg", "glu", "Y")
write.csv(db,'diabetes.csv')Vistazo a los datos
dt <- datatable(db)
# widgetframe::frameWidget(dt)
dtHay un problema con el uso de la librería
datatabley por eso no pinta la tabla. Con el paquetewidgetframese arregla pero desorganiza la página (deja mucho espacio tras presentar la tabla). Esa es la solución secundaría comentada en este hilo destackoverflow. Me falta probar la primera. + info
Y un sumario extendido vía la función dfSummary de summarytools
dfSummary(db)Data Frame Summary
db
Dimensions: 442 x 11
Duplicates: 0
| No | Variable | Stats / Values | Freqs (% of Valid) | Graph | Valid | Missing |
|---|---|---|---|---|---|---|
| 1 | Edad [integer] |
Mean (sd) : 48.5 (13.1) min < med < max: 19 < 50 < 79 IQR (CV) : 20.8 (0.3) |
58 distinct values | : . : : : : : : : . . . : : : : : : : : : : : : : : : . |
442 (100%) |
0 (0%) |
| 2 | Sexo [integer] |
Min : 1 Mean : 1.5 Max : 2 |
1 : 235 (53.2%) 2 : 207 (46.8%) |
IIIIIIIIII IIIIIIIII |
442 (100%) |
0 (0%) |
| 3 | IMC [numeric] |
Mean (sd) : 26.4 (4.4) min < med < max: 18 < 25.7 < 42.2 IQR (CV) : 6.1 (0.2) |
163 distinct values | : . . : : . : : : : . . : : : : : : : : : : : : . . |
442 (100%) |
0 (0%) |
| 4 | PAM [numeric] |
Mean (sd) : 94.6 (13.8) min < med < max: 62 < 93 < 133 IQR (CV) : 21 (0.1) |
100 distinct values | : : : : : : : : : : : . : : : : : . |
442 (100%) |
0 (0%) |
| 5 | tc [integer] |
Mean (sd) : 189.1 (34.6) min < med < max: 97 < 186 < 301 IQR (CV) : 45.5 (0.2) |
141 distinct values | : . : . : : : : : : : : : : : : : : : . . |
442 (100%) |
0 (0%) |
| 6 | ldl [numeric] |
Mean (sd) : 115.4 (30.4) min < med < max: 41.6 < 113 < 242.4 IQR (CV) : 38.5 (0.3) |
302 distinct values | : : : : : : : . : : : : : . : : : : : : . |
442 (100%) |
0 (0%) |
| 7 | hdl [numeric] |
Mean (sd) : 49.8 (12.9) min < med < max: 22 < 48 < 99 IQR (CV) : 17.5 (0.3) |
63 distinct values | : . : . : : : : : : : . : : : : . |
442 (100%) |
0 (0%) |
| 8 | tch [numeric] |
Mean (sd) : 4.1 (1.3) min < med < max: 2 < 4 < 9.1 IQR (CV) : 2 (0.3) |
66 distinct values | : : . : : . : : : : : : : . |
442 (100%) |
0 (0%) |
| 9 | ltg [numeric] |
Mean (sd) : 4.6 (0.5) min < med < max: 3.3 < 4.6 < 6.1 IQR (CV) : 0.7 (0.1) |
184 distinct values | : : : : : : : . : : : : : : : . |
442 (100%) |
0 (0%) |
| 10 | glu [integer] |
Mean (sd) : 91.3 (11.5) min < med < max: 58 < 91 < 124 IQR (CV) : 14.8 (0.1) |
56 distinct values | : : . : : : : : : . : : : : . . : : : : : : : . |
442 (100%) |
0 (0%) |
| 11 | Y [integer] |
Mean (sd) : 152.1 (77.1) min < med < max: 25 < 140.5 < 346 IQR (CV) : 124.5 (0.5) |
214 distinct values | : : . : : : : : : : : : : : : : : . |
442 (100%) |
0 (0%) |
Gráficos
model <- lm(
formula = Y ~ Edad + Sexo + IMC + PAM + glu + Sexo:IMC +
Sexo:PAM + IMC:PAM + Sexo:glu + IMC:glu + PAM:glu +
Sexo:IMC:PAM + Sexo:IMC:glu + Sexo:PAM:glu +
IMC:PAM:glu + Sexo:IMC:PAM:glu, data = db
)
p1 <- ggplot(model, aes(x = Edad, y = Y)) +
geom_point(aes(colour = factor(Sexo)), alpha = 0.4) +
stat_smooth(method = "lm", col = "blue")
p2 <- ggplot(model, aes(x = IMC, y = Y)) +
geom_point(aes(colour = factor(Sexo)), alpha = 0.4) +
stat_smooth(method = "lm", col = "blue")
p3 <- ggplot(model, aes(x = PAM, y = Y)) +
geom_point(aes(colour = factor(Sexo)), alpha = 0.4) +
stat_smooth(method = "lm", col = "blue")
p4 <- ggplot(model, aes(x = glu, y = Y)) +
geom_point(aes(colour = factor(Sexo)), alpha = 0.4) +
stat_smooth(method = "lm", col = "blue")
#grid.arrange(p1, p2, p3, p4, nrow = 2)
ggarrange(p1, p2, p3, p4, ncol = 2, nrow = 2, common.legend = TRUE, legend = "bottom")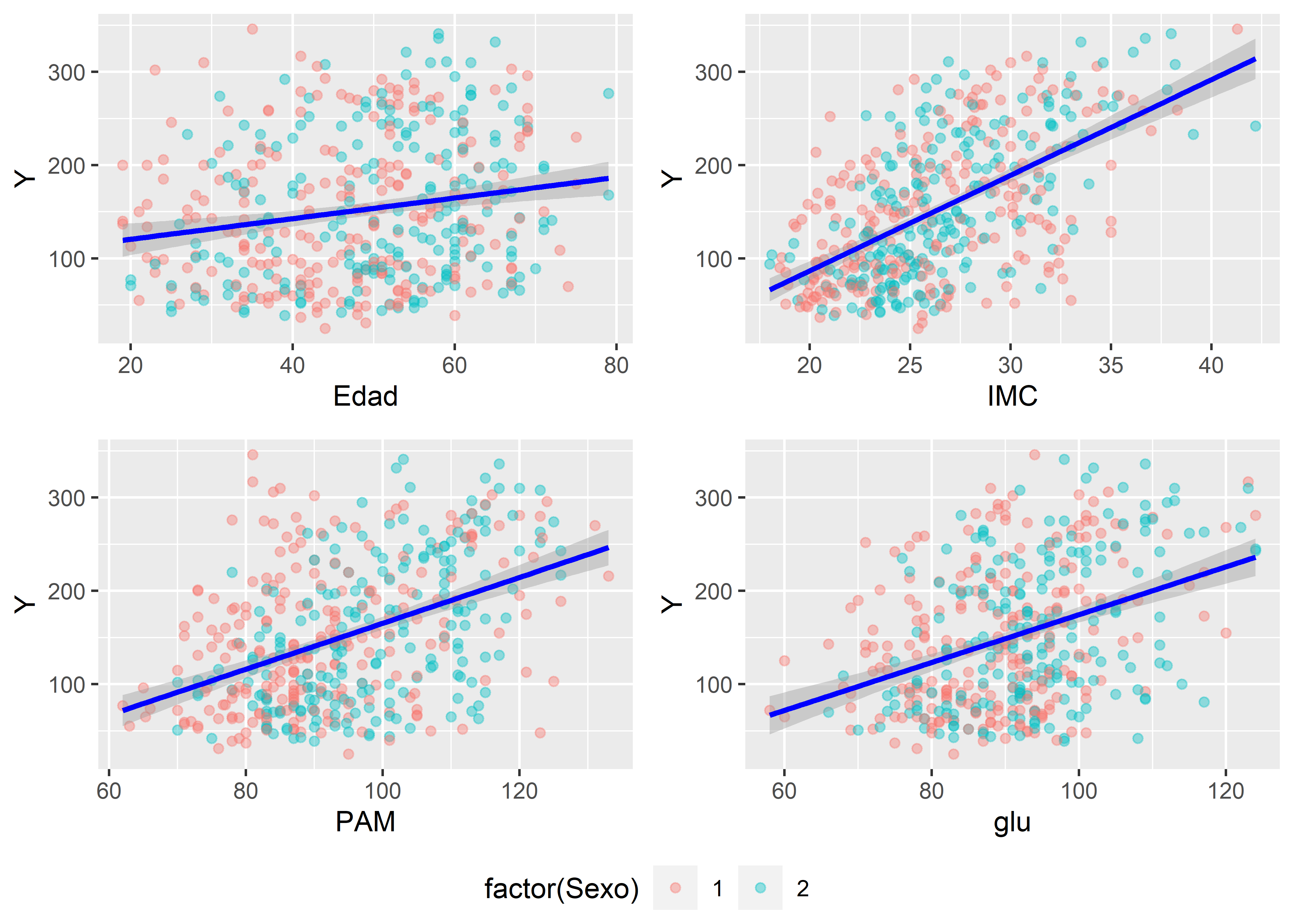
Consultas SQL
Algunos ejemplos de uso.
sql1 <- sqldf('select * from db where Edad > 50 and Edad < 54')
datatable(sql1)dfSummary(sql1)Data Frame Summary
sql1
Dimensions: 49 x 11
Duplicates: 0
| No | Variable | Stats / Values | Freqs (% of Valid) | Graph | Valid | Missing |
|---|---|---|---|---|---|---|
| 1 | Edad [integer] |
Mean (sd) : 52.1 (0.9) min < med < max: 51 < 52 < 53 IQR (CV) : 2 (0) |
51 : 16 (32.6%) 52 : 14 (28.6%) 53 : 19 (38.8%) |
IIIIII IIIII IIIIIII |
49 (100%) |
0 (0%) |
| 2 | Sexo [integer] |
Min : 1 Mean : 1.4 Max : 2 |
1 : 30 (61.2%) 2 : 19 (38.8%) |
IIIIIIIIIIII IIIIIII |
49 (100%) |
0 (0%) |
| 3 | IMC [numeric] |
Mean (sd) : 26.2 (3.2) min < med < max: 19.7 < 26.4 < 33.2 IQR (CV) : 4.3 (0.1) |
40 distinct values | : : : : : : : : : : : : : . : : : : : : : |
49 (100%) |
0 (0%) |
| 4 | PAM [numeric] |
Mean (sd) : 95.5 (11.2) min < med < max: 76 < 94 < 117 IQR (CV) : 17.3 (0.1) |
36 distinct values | : . : : : : : : : : : : : : : : : : : : : : : : : : : : . |
49 (100%) |
0 (0%) |
| 5 | tc [integer] |
Mean (sd) : 192.5 (32.3) min < med < max: 119 < 195 < 280 IQR (CV) : 39 (0.2) |
41 distinct values | : : . : : : . : : : . . . : : : : : . |
49 (100%) |
0 (0%) |
| 6 | ldl [numeric] |
Mean (sd) : 117.2 (29.4) min < med < max: 53.4 < 119 < 175.6 IQR (CV) : 36.4 (0.3) |
47 distinct values | . : : : : : : . : : : : : : : : : : : : |
49 (100%) |
0 (0%) |
| 7 | hdl [numeric] |
Mean (sd) : 51.4 (13.8) min < med < max: 29 < 48 < 93 IQR (CV) : 18 (0.3) |
32 distinct values | : : : : : : : . : : : : . . . |
49 (100%) |
0 (0%) |
| 8 | tch [numeric] |
Mean (sd) : 4 (1.3) min < med < max: 2 < 4 < 8 IQR (CV) : 2 (0.3) |
15 distinct values | : : : : : : : : : : : : : . . |
49 (100%) |
0 (0%) |
| 9 | ltg [numeric] |
Mean (sd) : 4.7 (0.4) min < med < max: 3.9 < 4.7 < 5.8 IQR (CV) : 0.7 (0.1) |
43 distinct values | . . . : : : : : : : : : : : : . : : : : : : : : : : : : : : : : . . |
49 (100%) |
0 (0%) |
| 10 | glu [integer] |
Mean (sd) : 92.7 (10.6) min < med < max: 69 < 93 < 120 IQR (CV) : 14 (0.1) |
28 distinct values | : . : : : : : : : : : : . |
49 (100%) |
0 (0%) |
| 11 | Y [integer] |
Mean (sd) : 160.9 (82.2) min < med < max: 44 < 154 < 292 IQR (CV) : 157 (0.5) |
46 distinct values | : : . : : . : . : . : : : : : : : : : : : |
49 (100%) |
0 (0%) |